Transcripción
en eucariotas
En el caso de las eucariotas, el proceso se realiza en el núcleo,
y es similar al de las procariotas, pero de mayor complejidad. Diferentes ARNp
transcriben distintos tipos de genes. La ARNpII transcribe los pre-ARNm,
mientras que la ARNpI y ARNpIII transcriben los ARN-ribosomales y ARNt,
respectivamente. Los ARNs transcritos son modificados posteriormente. El
pre-ARNm,por ejemplo, sufre un proceso de maduración que tras cortes y empalmes
sucesivos elimina ciertos segmentos del ADN llamados los intrones para
producir el ARNm final. Durante este proceso de maduración se puede dar lugar a
diferentes moléculas de ARN, en función de diversos reguladores. Así pues, un
mismo gen o secuencia de ADN, puede dar lugar a diferentes moléculas de ARNm y
por tanto, producir diferentes proteínas. Otro factor de regulación propio de
las células eucariotas son los conocidos potenciadores (en inglés: "enhancers"),
que incrementan mucho (100 veces) la actividad de transcripción de un gen, y no
depende de la ubicación de éstos en el gen, ni la dirección de la lectura.
Etapas de la transcripción
Clásicamente se divide el proceso de la transcripción
en 3 etapas principales (iniciación, elongación y terminación), pero realmente
se pueden diferenciar 5 etapas :
Preiniciación
Al contrario de la replicación de ADN, durante el inicio de la
transcripción no se requiere la presencia de un cebador para sintetizar la
nueva cadena, de ARN en este caso. Antes del inicio de la transcripción se
necesitan toda una serie de factores de transcripción que ejercen
los factores de iniciación. Estos se unen a secuencias específicas de ADN para
reconocer el sitio donde la transcripción ha de comenzar y se sintetice el ARN
cebador. Esta secuencia de ADN en la que se ensamblan los complejos de
transcripción se llama promotor.
Los promotores se localizan en los extremos 5'-terminales de los genes, antes del comienzo
del gen, y a ellos se unen los factores de transcripción mediante fuerzas de Van der Waals yenlaces de hidrógeno. Los promotores
tienen secuencias reguladoras definidas, muy
conservadas en cada especie, donde las más conocidas son la caja TATA (situada
sobre la región -10), con la secuencia consenso TATA(A/T)A(A/T); y la caja
TTGACA (situada en el punto -35). La formación del complejo de transcripción se
realiza sobre el promotor TATA, allí se forma el núcleo del complejo de
iniciación. Sobre la caja TATA se fija una proteína de unión (TBP) junto con el
factor de transcripción TFII D (TF proviene del inglés: transcription
factor). Después, a ellos se unen otros factores de transcripción
específicos: TFII A, que estabiliza el complejo TFII D-ADN;
los factores TFII B y TFII E se unen al ADN y
el TFII F (una helicasadependiente
de ATP) y al final la ARN polimerasa. Todo ello
forma un complejo que se llama complejo de preiniciación cerrado.
Cuando la estructura se abre por mediación del factor de transcripción TFII
H, da comienzo la iniciación..
Iniciación
Primero, una Helicasa separa las hebras de ADN en
estas denominadas cajas TATA, ya que entre adenina y timina se establecen dos
enlaces de hidrógeno, mientras que entre citosina y guanina se forman tres.
Posteriormente se unen los factores y las proteinas de transcripción (TBP,
TF2D, TF2B) permitiendo, de esta manera, el acceso de la ARN polimerasa al
molde de ADN de cadena simple, siendo esta la ultima en posicionarse. Aunque la
búsqueda del promotor por la ARN polimerasa es muy rápida, la formación de la burbuja
de transcripción o apertura del ADN y la síntesis del cebador es muy
lenta. La burbuja de transcripción es una apertura de ADN desnaturalizado de 18
pares de bases, donde empieza a sintetizarse el ARN cebador a partir del
nucleótido número 10 del ADN molde de la burbuja de transcripción. La burbuja
de transcripción se llama complejo abierto. La ARN polimerasa es
una enzima formada por 5 subunidades: 2 subunidades α, 1 subunidad β, 1
subunidad β' y 1 subunidad ω que tiene como función la unión de ribonucleótidos trifosfato.
Cuando se forma el complejo abierto, la ARN polimerasa comienza a unir ribonucleótidos
mediante enlaces fosfodiéster, y una vez que se forma el primer
enlace fosfodiéster, acaba la etapa de iniciación.y comienza asi la siguiente
etapa.
Disgregación
del promotor
Una vez sintetizado el primer enlace fosfodiéster, se
debe deshacer el complejo del promotor para que quede limpio para volver a
funcionar de nuevo. Durante esta fase hay una tendencia a desprenderse el
transcrito inicial de ARN y producir transcritos truncados, dando lugar a una
iniciación abortada, común tanto en procariontes como eucariontes. Una vez que
la cadena transcrita alcanza una longitud de unos 23 nucleótidos, el complejo
ya no se desliza y da lugar a la siguiente fase, la elongación.
La disgregación del promotor coincide con una
fosforilación de la serina 5 del dominio carboxilo terminal de la ARN
polimerasa, que es fosforilado por el TFII H (que es una
proteínaquinasa dependiente
de ATP)
Elongación
La ARN polimerasa cataliza la elongación de cadena del
ARN. Una cadena de ARN se une por apareamiento de bases a la cadena de ADN, y
para que se formen correctamente los enlaces de hidrógeno que determina el
siguiente nucleótido del molde de ADN, el centro activo de la ARN polimerasa
reconoce a los ribonucleótidos trifosfato entrantes. Cuando el nucleótido
entrante forma los enlaces de hidrógeno idóneos, entonces la ARN polimerasa
cataliza la formación del enlace fosfodiéster que corresponde. A
esto se le llama elongación, la segunda etapa de la transcripción del ARN.
Terminación
Al finalizar la síntesis de ARNm, esta molécula ya se
ha separado completamente del ADN (que recupera su forma original) y también de
la ARN polimerasa, terminando la transcripción. La terminación es otra etapa
distinta de la transcripción, porque justo cuando el complejo de transcripción
se ha ensamblado activamente debe desensamblarse una vez que la elongación se
ha completado. La terminación está señalizada por la información contenida en
sitios de la secuencia del ADN que se está transcribiendo, por lo que la ARN
polimerasa se detiene al transcribir algunas secuencias especiales del ADN.
Estas secuencias son ricas en guanina y citosina,
situadas en el extremo de los genes, seguidas de secuencias ricas en timina,
formando secuencias palindrómicas,
que cuando se transcriben el ARN recién sintetizado adopta una estructura
en horquilla que desestabiliza el complejo ARN-ADN, obligando a
separarse de la ARN polimerasa, renaturalizándose la burbuja de transcripción.
Algunas secuencias de ADN carecen de la secuencia de terminación, sino que
poseen otra secuencia a la que se unen una serie de proteínas reguladoras
específicas de la terminación de la transcripción como rho
ARNm EUCARIOTA
1- La mayoría de los ARNm eucariotas presentan la secuencia del mensaje
interrumpida, es decir coexisten a lo largo de la molécula sectores que codifican para
la proteína llamados EXONES, con secuencias intercaladas sin información
denominadas
INTRONES (Fig. 12.9).
Fig.
11.9 - Estructura en mosaico del
ARNm transcripto primario eucariota
2- Los ARNm transcriptos primarios sufren una serie de modificaciones antes de salir al citoplasma como ARNm maduro.
Algunos cambios consisten en
el agregado de moléculas en los extremos 5' y 3' llamados capping y poliadenilación.
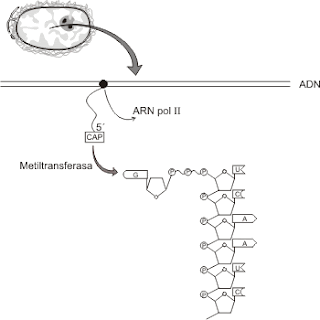
Capping: Ésta es la denominación
del proceso por el cual se adiciona en el extremo 5' del ARNm una molécula de 7
metil-guanosina (un nucleótido metilado) a la que se conoce como cap (del inglés, capuchón). Ésta molécula se agrega al
ARNm naciente cuando éste alcanza, aproximadamente, los 30 nucleótidos de
longitud. Por ser esta modificación simultánea a la transcripción se la
considera co-transcripcional. La cap impide la degradación del ARNm inmaduro
por nucleasas y fosfatasas nucleares. También participa en la remoción de
intrones y en el inicio de la traducción.
Fig
11.10- El agregado del cap
Poliadenilación: Este proceso consiste en el agregado de alrededor de
unas 250 adenosinas
o cola poli A , en el extremo
3' del ARNm. El ARNm presenta una secuencia de
nucleótidos
específica AAUAAA conocida
como señal de poliadenilación. Una nucleasa corta
al pre-ARNm a unos 20
nucleótidos después de la señal. Una vez liberado el pre-ARNm, la enzima
poliA-polimerasa le agrega las adenosinas de a una por vez. Por otra parte la
ARN pol II continúa transcribiendo un tramo más del molde, para finalmente
disociarse del gen. Este último tramo de ARN es totalmente infructuoso, pues
resulta rápidamente degradado por nucleasas y fosfatasas.
Las funciones de la cola
poli A son: proteger el extremo 3' de la degradación y ayudar a los ARNm a
salir del núcleo.
Carecen
de cola poli A los ARNm de histonas, dado que como ya se mencionó, no presentan
la señal de poliadenilación. Otra excepción la ejemplifican algunos genes que
presentan más de una señal de poliadenilación. Cuando así ocurre, el mismo gen
puede ser transcripto en dos productos diferentes, dependiendo de cuál sea la
señal reconocida.
Fig.
11.11 - El agregado de la cola poli A
Otra modificación significativa afecta a la secuencia codificadora, que sufre un acortamiento producto de la eliminación de los intrones, quedando como producto final los exones empalmados en secuencia continua. Este tipo de procesamiento se llama empalme (splicing) y fue descubierto en 1977.
Para
que se lleve a cabo este tipo de maduración del pre-ARNm se necesita una
batería de ribonucleoproteínas nucleares: las
RNPpn ( snRNP, del inglés, small nuclear ribonucleoprotein).
Estas partículas ricas en uridinas y diversas proteínas se denominan U1,
U2, U4, U5, U6 y
se combinan de a una por vez en los extremos de cada intrón (lindantes a sendos
exones). El complejo resultante del ensamblado de las distintas RPNpn se
denomina espliceosoma.
La actividad enzimática residiría en los ARNpn del espliceosoma. Éstos serían
los responsables de reconocer las secuencias señalizadoras de corte, escindir a
los intrones y empalmar a los exones entre ellos, produciendo una molécula de
ARNm maduro.
Mecanismo molecular del
splicing
El corte de intrones y el empalme
de exones deben ser muy exacto, pues de lo contrario un error pequeño, causaría
un corrimiento del marco de lectura del ARNm. Los intrones son clivados del
transcripto primario por las RNPpn que reconocen cortas secuencias conservadas
dentro del intrón y en los límites con los exones. Estas secuencias, muy
similares en todos los intrones estudiados, son:
· Secuencia GU en el extremo 5' o sitio dador
· Secuencia AG en el extremo 3' del intrón
o sitio aceptor
· Secuencias conocida como sitio de ramificación que se
localiza en el interior del intrón
Estas secuencias participan
en las reacciones de clivado y empalme que ocurren en dos etapas:
En la primer etapa, una
RNPpn reconoce al sitio dador y otra se enlaza al sitio de ramificación. El
extremo 5' es clivado y ligado a un nucleótido(A) del sitio de ramificación.
En la segunda etapa, se da
el reconocimiento del extremo 3’ por parte de otra RNPpn y se produce el corte
en el extremo 3' del intrón, seguido por el empalme de los dos exones. Así se
libera el ARNm maduro del espliceosoma. El intrón queda eliminado en forma de
lazo y será degradado posteriormente en el núcleo.
Fig.
11.12 - Acción del espliceosoma
Como citáramos anteriormente, la presencia del capuchón en el extremo 5' es requerida para que las RNPpn trabajen, no así la cola poli A.
3- Los ARNm maduros
son monocistrónicos, es decir el sector
codificador dicta la secuencia para una sola cadena polipeptídica.
Fig. 11.13 - Estructura de la molécula de ARNm maduro eucariota
ARNm PROCARIOTA
1-Los ARNm procariotas
presentan las secuencias codificadoras continuas, pues carecen de
intrones.
2-No sufren modificaciones
post-transcripcionales.
3-Muchos ARNm procariotas son policistrónicos, es decir que una sola molécula de ARNm contiene
información para varias proteínas. Este ARNm contiene codones para la
terminación y para la iniciación de la traducción entre las secciones
codificadoras de proteínas, de modo que se traduce como varias moléculas de
proteína distintas.
Fig. 11.14 - Estructura de la molécula de ARNm procariota
ARNt (TRANSFERENCIA)
Los ARNt son moléculas
"adaptadoras" ya que interactúan por un lado con la cadena
polinucleotídica (ARNm) y por el otro con los aminoácidos que formarán parte de
la cadena polipeptídica, así alinean a los aminoácidos siguiendo el orden de
los codones del ARNm.
Cada ARN t es una molécula de
70 a 93 ribonucléotidos. Enlaces puente hidrógeno entre sus bases repliegan la
molécula dando origen a una disposición espacial en forma de trébol (Fig.11.15).
Cada brazo presenta una zona de doble cadena y un asa o bucle de cadena simple
sin apareamiento de bases. Interacciones posteriores doblan la estructura de
trébol formando una "ele".
¿Por qué existen 64 codones y aproximadamente 31
ARNt, si en la naturaleza hay 20 aminoácidos ?
Existen 64 combinaciones
posibles en que las 4 bases pueden ordenarse en tripletes o codones. De ellos,
3 son codones stop. Los 61 tipos restantes codifican para los 20 aminoácidos,
existiendo para varios de ellos codones sinónimos (ver tabla 11.2).
Los anticodones de los ARNt
reconocen a los codones del ARNm, sin embargo no hay 61 tipos distintos de ARNt
porque no se requiere la complementaridad exacta entre los 3 nucleótidos del
codón y el anticodón.
En muchos casos, mientras
coincidan las 2 primeras bases del codón, hay suficiente "balanceo"en
la tercera posición para permitir el acoplamiento con más de un tipo de
nucleótido en el anticodón, de manera que existen aproximadamente 31 tipos de
ARNt.
Por
ejemplo el aminoácido fenilalanina es codificado por dos codones sinónimo: UUU
/ UUC. Sin embargo un solo anticodón AAG puede
complementarse indistintamente con UUU y UUC, realizando el trabajo de llevar a la fenilalanina al
ribosoma para ser incorporada a la cadena polipeptídica en formación .
Todos los ARNt presentan un porcentaje significativo de bases poco frecuentes que surgen por modificación post-transcripcional de los cuatro ribonucleótidos estándar A, G, C y U (Fig. 11.16).
Fig. 11.15 - Estructura del ARNt
Fig. 11.16 - Bases raras en el ARNt
En esta molécula se distinguen
básicamente dos extremos:
En el extremo 3' o extremo aceptor de la molécula se encuentra el
trinucleótido CCA que representa el sitio de unión donde se liga el aminoácido.
Esta unión es catalizada por una enzima aminoacil ARNt sintetasa específica y apropiada para
cada uno de los 20 aminoácidos.
En el otro extremo de la
"ele" se localiza un triplete de nucleótidos conocido como anticodón, cuya secuencia varía en cada
tipo de ARNt. Cada anticodón es complementario de un codón del ARNm, aunque
podría acoplarse a más de un codón (sinónimo).
Por lo hasta aquí expuesto,
podemos concluir que el ARNt debe poseer varias propiedades específicas:
· Debe ser reconocido por una aminoacil ARNt sintetasa
que lo una al aminoácido correcto.
· Debe tener una región que actúe como sitio de unión
para el aminoácido.
· Debe tener una secuencia complementaria (anticodón)
específica para el codón del ARNm correcto.
Las modificaciones que
sufren los pre-ARNt son semejantes en procariotas y en eucariotas en cuanto en
ambos se producen escisiones y modificaciones químicas, por ejemplo se elimina
un dinucleótido 3' del precursor y se agrega el triplete CCA a los ARNt que no
poseen todavía esta secuencia terminal. También aparecen bases raras
(Fig.11.16) por modificación enzimática de nucleótidos estándar del ARNt
precursor.
Algunos genes para ARNt
presentan un intrón que interrumpe la secuencia codificadora, él cual es
removido post-transcripción. (Fig.11.17)
Fig. 11.17 - Intrón en el ARNt
ARNr (RIBOSÓMICO)
Los
ARNr, junto a proteínas, son los componentes de los RIBOSOMAS. Los ribosomas y
los ARNt intervienen en la traducción de la información codificada en el ARNm
por lo cual los primeros son considerados fábricas
de proteínas.
Cada ribosoma consta de dos
subunidades: la subunidad MAYOR y la subunidad MENOR (Fig. 11.18)
Fig. 11.18 - Modelo tridimensional del ribosoma bacteriano visto desde distintos ángulos
La subunidad mayor contiene
una depresión en una de sus superficies, en la cual se ajusta la subunidad
menor. El ARNm se inserta en el surco formado entre las superficies de contacto
de las subunidades. (Fig. 11.19).
Dentro de cada ribosoma hay
dos huecos llamados sitios A (AMINOACÍDICO) y P (PEPTIDÍLICO), para el ingreso
de los ARNt unidos a sus correspondientes aminoácidos. (Fig. 11.20)
Fig. 11.19- Modelo de ribosoma que representa la ubicación tentativa del ARNm y de la proteína naciente
Fig. 11.20- Sitios aminoacídico y peptidílico del ribosoma
Las subunidades ribosómicas
trabajan conjuntamente para la síntesis proteica. La subunidad menor aloja al
ARNm sobre el cual se acomodan los ARNt para que puedan unirse los aminoácidos
que transportan.
La
subunidad mayor cataliza la unión peptídica de los aminoácidos gracias a la
acción de la peptidil transferasa ( enzima que forma parte
de la estructura de esta subunidad). [1]
Si
bien cumplen idéntica función, los ribosomas procariotas y eucariotas se
diferencian en su tamaño, coeficiente de sedimentación [2] , y en
el tipo y número de moléculas de ARNr y proteínas que los forman:
Fig.
11.21- Comparación de las estructuraas de los ribosomas procariotas y
eucariotas
Todos los ARNr procariotas y
eucariotas sufren modificaciones post-transcripción.
En eucariotas los ARNr 18S;
5,8S y 28S son transcriptos de un mismo gen que codifica para un pre-ARNr 45S.
La síntesis del este transcripto primario se realiza en la zona fibrilar del
nucléolo a partir de la ARN pol I. Una vez obtenido el largo transcripto
primario, se eliminan las secuencias espaciadoras(tramos de ARN inútiles para
integrar la estructura ribosómica), las que serán degradadas por enzimas. Las
secuencias resultantes utilizables de ARNr (28S, 18S y 5,8S) son metiladas y
junto con ARNr 5S extranucleolar, se ensamblan a proteínas importadas del
citoplasma para conformar la subunidades ribosómicas (Fig. 11.22).
Fig. 11.22 - Procesamiento de los ARN 45S
Las subunidades ribosómicas
en distintos estadios de ensamblaje, se localizan en la periferia del nucléolo,
conformando la zona granular del mismo.
Finalmente, las subunidades
ribosómicas por separado y antes de completar sus respectivos ensambles, son
exportadas al citoplasma a través del complejo del poro, concluyendo el
procesamiento de cada subunidad en el citosol.
Respecto del ARNr 5S no
transcripto en el nucléolo, una vez sintetizado se incorpora al resto de los
componentes para conformar la subunidad mayor del ribosoma (Fig.11.23).
Fig. 11.23 - Ensamblaje de subunidades ribosómicas
LOS ARN PEQUEÑOS
Los ARN pequeños forman
complejos con proteínas específicas dando lugar a la formación de partículas
ribonucleoproteicas (RNP).
Las
RNP localizadas en el núcleo se denominan RNPpn: partícula ribonucleoproteica pequeña ( sn RNP/s:
small -pequeña- /n: nuclear-RNP:ribonucleoproteica). Varios RNPpn conocidos
como U1 a U6 participan en el procesamiento de los ARNm. Estas partículas
forman un complejo multienzimático denominado espliceosoma, encargado de realizar cortes y empalmes en los ARNm
transcritos primarios (splicing) .
Las
RNP localizadas en citoplasma se denominan RNPpc: partícula ribonucleoproteica pequeña citoplasmática
(scRNP/s: small/ c:cytosolic-citosol-/RNP:ribonucleoproteica). La función más
conocida de cualquier RNPpc es la que desempeña el complejo ARN /proteínas que
componen la partícula dereconocimiento de la señal o SRP. Estas partículas participan en el reconocimiento de
secuencias específicas de las proteínas de secreción, membranares y
lisosomales, deteniendo su traducción hasta que el ribosoma en donde se están
sintetizando se contacte con la membrana del retículo endoplasmático granular,
en donde finalizan su traducción.
bibliografia:
www.genomasur.com/lecturas/Guia11.htm

















No hay comentarios:
Publicar un comentario